Практикум 4
Задание 1
В результате работы сайта RNAfold я получил две вторичные структуры тРНК(рис 2).На тРНК больше похожа структура слева.В результате работы алгоритма Зукера получилась структура не похожая на тРНК.
| Участок структуры (расшифровку названий | Позиции в структуре (по результатам find_pair по номерам остатков) | Результаты предсказания с помощью сайта RNAfold | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 901-907 и 972-966 | 6 | 5 |
| D-стебель | 910-913 и 922-925 | 6 | 7 |
| T-стебель | 949-953 и 961-965 | 5 | 7 |
| Антикодоновый стебель | 939-944 и 926-931 | 5 | 5 |
| Общее число канонических пар нуклеотидов | 18 | 22 | 24 |
Рис 1.Результат работы алгоритма Зукера
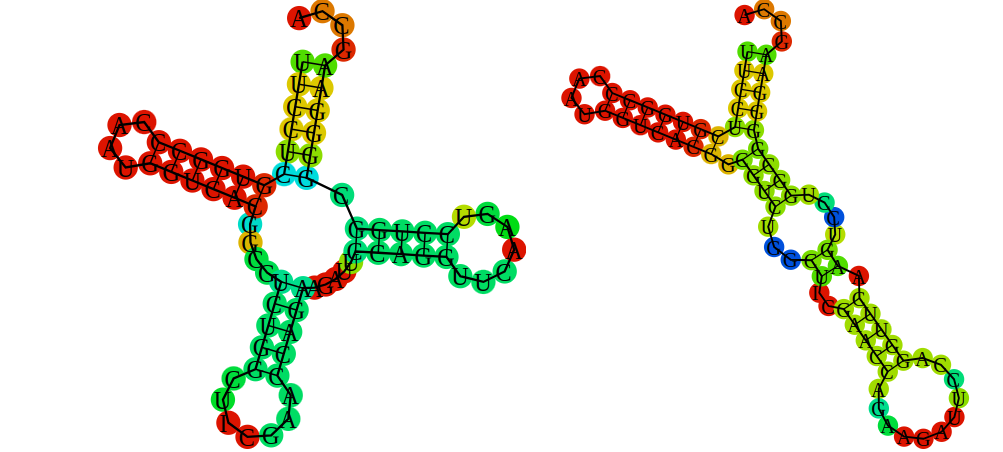 Рис 2.Результат работы сервиса RNAfold
Рис 2.Результат работы сервиса RNAfold
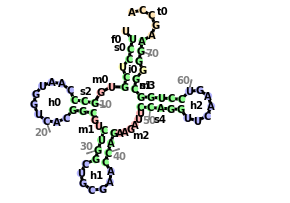 Рис 3.Результат работы forgi
Рис 3.Результат работы forgi
Задание 2
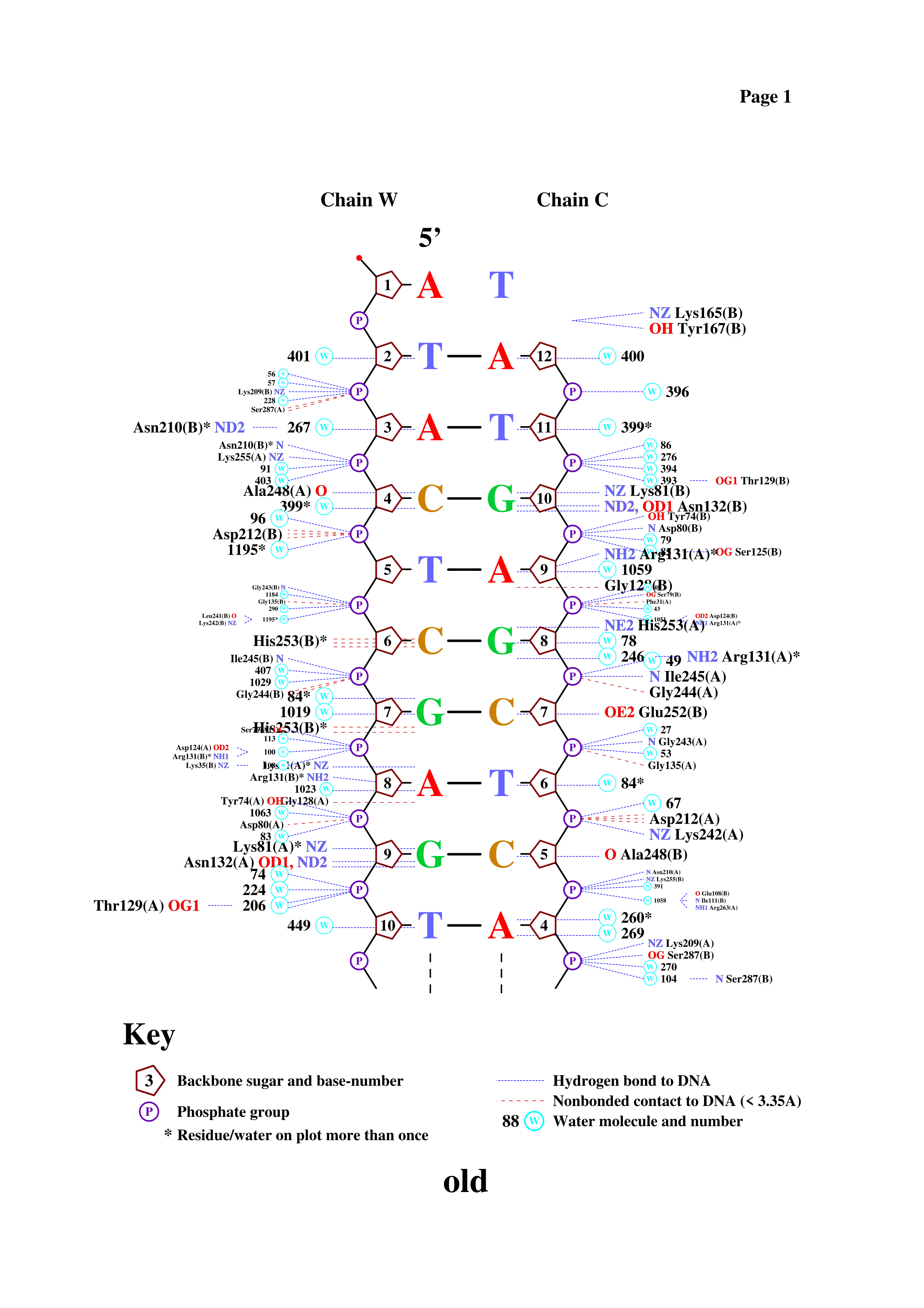
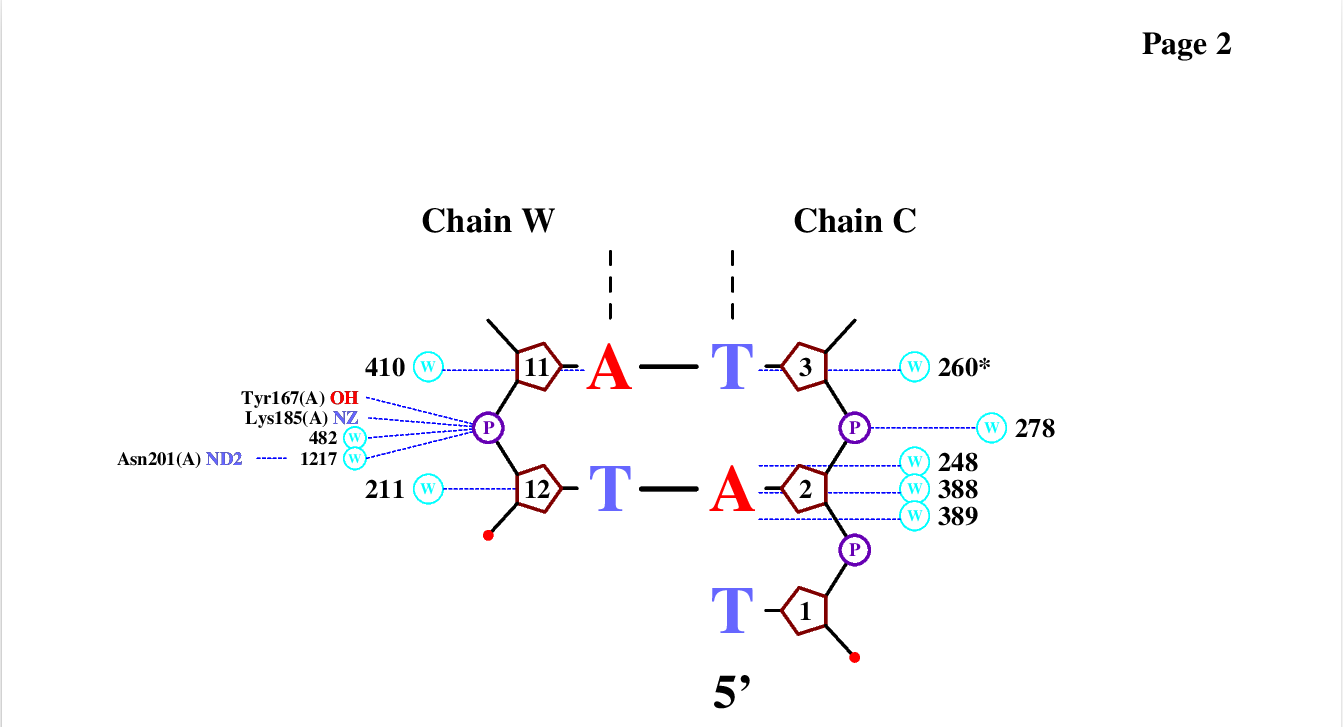
Я буду работать в Pymol.Кислороды соединяющие атомы фосфора и остовы сахара считаем принадлежащими сахару.
Скрипт Pymol с выделением 3-х групп.
Скрипт с различными отображениями После его активации в левом нижнем углу появятся 3 вкладки,
каждая из которых переключает на отображение соответствующие заданию.
Для заполнения таблицы помог этот скрипт.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 39 | 43 |
| остатками фосфорной кислоты | 34 | 19 | 53 |
| остатками азотистых оснований со стороны большой бороздки | 11 | 20 | 31 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 4 | 4 |
nucplot выдает ошибку на мою структуру 1TRO и не создает файл .ps(да я переводил pdb в старый формат)
No bond file supplied:-
Reading external interaction information from PDB CONNECT records
and HBPLUS output files
Segmentation fault.
Поэтому я вместо первой сверху структуры взял вторую сверху(1dc1).
Наиболее связан с ДНК Lys81B он и наиболее важен для распознования ДНК